迄今为止,最大的人工合成基因组已经完成,其氨基酸编码密码子的数量比通常要少。
这提高了编码含有非自然氨基酸残基的蛋白质的可能性。
图1 大肠杆菌的通用图片,研究人员重新设计了该生物的遗传密码
在过去的10年中,化学合成DNA成本的降低和DNA片段组装方法的改进使得科研人员能够将合成生物学的规模扩大到产生整个染色体和基因组的水平。之前,合成DNA已经构建了多达100万碱基对,尤其是一组来自酿酒酵母的染色体和支原体分枝杆菌的多个版本的基因组。现在,朱利叶斯•福莱顿斯(Julius Fredens)等人在《自然》杂志上发表文章,报道了一个有着400万碱基对的大肠杆菌基因组合成版本的完成。剑桥大学分子生物学家贾森•秦(Jason Chin)是这篇论文的通信作者。这是合成基因组学新兴领域的一个里程碑,并最终将该技术应用于实验室的主力细菌。
合成基因组学为理解生命规律提供了一种新方法,同时也将合成生物学推向了一个可以编写基因组进行设计的未来。该领域的先驱——马里兰州罗克维尔市克雷格•文特尔研究所的科研人员——已经使用这种方法更好地定义了独立生存的细胞所需的最小基因集。通过采用计算机重新设计基因组片段、化学合成片段并进行组装的方法,先驱者们成功地将分枝杆菌基因组的大小缩减了大约50%。根据以往对大肠杆菌的研究表明,仅仅使用基因组编辑工具进行同样的操作将会更加费力:基因删除法最多只能删除基因组的15%。
福莱顿斯和他的同事们利用大肠杆菌的这种简化版基因组作为合成基因组的模板,同时也考虑了另一种最小化——密码子简化。遗传密码存在固有的冗余:共有64个密码子(3个一组的“字母”或碱基),却只能编码20个氨基酸,再加上标记蛋白质编码序列开始和结束的起始密码子和终止密码子。这种冗余意味着,例如,有6个编码丝氨酸的密码子,以及3个可能的终止密码子。通过设计、合成和组装,福莱顿斯等人能够在其编码蛋白质的序列中只使用64个密码子中的61个,来构造一个大肠杆菌基因组。他们用同义密码子(“拼写”不同,但是能给出相同翻译指令的密码子)替换掉了两个丝氨酸密码子和一个终止密码子。过去的工作中使用了基因组编辑工具已经产生了一种合成大肠杆菌,它只使用64个密码子中的63个,但这只需要将序列为TAG的终止密码子(基因组中只含有321个这种终止密码子)换成另一种终止密码子。减少到61个密码子则需要对多达18 214个密码子进行改变,这就必须用到基因组合成方法。
福莱顿斯和他的同事们利用大规模DNA组装和基因组整合的方法构建了他们的合成大肠杆菌基因组。他们先前已经利用这些方法对大肠杆菌中密码子更改极限进行了探索。在他们的方法中,DNA被计算机设计,化学合成并组装成大小为100千碱基的片段,并在酿酒酵母中将其装入载体;然后,这些载体被转入大肠杆菌中,并直接整合入基因组对应的自然区域中。将这一过程重复5次,结果就是DNA中的500千碱基片段被合成版本所取代。他们用这种方法生产了8株大肠杆菌,每一株都携带有合成的DNA片段,涵盖基因组的不同区域。然后用接合法将这些片段接合起来,形成完整的合成基因组。
这次大规模构建极其成功,脱靶突变率非常低,但也并非没有挑战性。大肠杆菌基因组中的许多基因与其他基因均有部分重叠,在91例重叠区域中均包含需要进行改变的密码子。这很复杂,因为一个蛋白质编码序列的同义变化有可能会引起重叠序列编码的氨基酸的改变。为了解决这个问题,研究小组“重构”了基因组中的79个位点,复制序列,将重叠的编码序列分离成单独的编码序列(图2)。
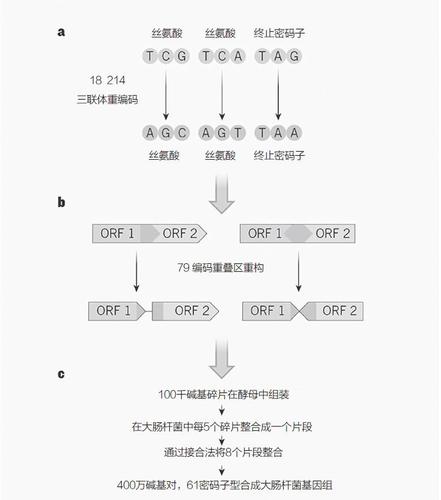
图2 重编码基因组的设计和构建
a·福莱顿斯等人重新编码了三个碱基三联体(密码子)——编码丝氨酸的TCG和TCA,以及一个标志着蛋白质编码序列结束的终止密码子TAG——在大肠杆菌基因组中将其替换为具有相同功能(AGC,AGT和TAA分别替换)的同义密码子。b·在一些基因组位点上,开放阅读框(ORFs;蛋白质编码区域)重叠,一个ORF中密码子的变化可能会给重叠区域带来有害变化。福莱顿斯等人“重构”了这些ORFs,将它们分离,如图中所示,ORF1和ORF2(左边的两个ORFs“读取”方向相同;右边的两个“读取”方向相反)。c·重新设计的DNA在酿酒酵母中合成并组装成100千碱基碎片,然后将碎片组合成片段并整合入大肠杆菌基因组中。这些片段结合在一起产生了完整的功能性合成基因组
最终的菌株被证明可以存活,并能够在一系列典型的实验室条件下生长,尽管与野生型相比活力略弱。它不再使用终止密码子TAG和两个丝氨酸密码子TCG和TCA,因此识别这些密码子的细胞装置现在就可以被删除或重新分配,用以募集大多数活细胞通常使用的20种氨基酸以外的“非典型”氨基酸。这种招募在63密码子型的大肠杆菌中已被证明是有用的,从生物技术项目角度来看,非典型氨基酸被编码到所需的序列位置,以提供能够参与自然蛋白质所不能参与的化学反应的残基;从生物安全角度来看,可读的DNA编码信息在合成大肠杆菌内和外的自然传输是有限的,因为细胞的遗传密码的运作方式与自然界其他生命体略有不同。预计所有这些用途都将在新的61密码子型大肠杆菌中得到扩展,该大肠杆菌有潜力编码使用多个非典型氨基酸,并生成一个更为严格的基因防火墙(因为64个密码子中有3个不再被识别)。
合成一个拥有400万碱基对的基因组,并将遗传密码减少到61个密码子,这是合成基因组学的新纪录,但这个新纪录可能不会维持太久。国际Sc2.0联盟即将合成全部16个染色体共1 200万碱基对的酿酒酵母基因组——真核生物首个合成基因组,真核生物包括植物、动物和真菌——57密码子型大肠杆菌基因组的合成工作也正在进行中。此外,科学家们还在构建一种比野生型少两个密码子的鼠伤寒沙门氏菌基因组。未来,这可能会使携带合成基因组的细菌能够作为基于细胞的技术应用于人类肠道。
从技术的角度来看,所有这些不同的项目的最有趣的方面是合成基因组的工作流结构非常相似——千碱基的合成DNA片段在酵母细胞中(通过同源重组的过程)被组装成50到100千碱基的碎片,之后(通过可选择的重组方法)这些碎片被用来取代靶标生物体内的自然序列。方法标准化将使步骤自动化,更多的研究小组将会进入该领域。基因组最小化和密码子简化只是这项新技术的第一个用途,终有一天这项新技术将会帮助我们构建出功能重组的基因组和被设计用来指导细胞执行特定任务的个性化定制基因组。

